TIANSeq HiFi Amplification Mix
Sa ilog
Ang amplification ng PC ng NGS library, 1st-henerasyon ng pagkakasunud-sunod ng paglaki ng PCR, pag-clone ng mataas na fidelity, pagtuklas ng SNP, pagbago ng tukoy sa site, atbp.
Mga Tampok
■ Paglaki ng mataas na kahusayan: Tiyaking ang rate ng conversion at bawasan ang mga siklo ng amplification.
■ Mababang Kagustuhan: Balanseng pagpapahusay ng pagpapahusay para sa mga template ng DNA na may iba't ibang mga nilalaman ng GC%.
■ Mataas na pagtutukoy: Sa pag-aari ng HotStart at malakas na detalye.
■ Mataas na katapatan: Ang katapatan ay 50 beses na mas mataas sa Taq DNA polymerase.
■ Mataas na pagiging sensitibo: Ang pag-input ng template ay maaaring maging mas mababa sa 1 pg.
Ang lahat ng mga produkto ay maaaring ipasadya para sa ODM / OEM. Para sa mga detalye,mangyaring i-click ang Customized Service (ODM / OEM)
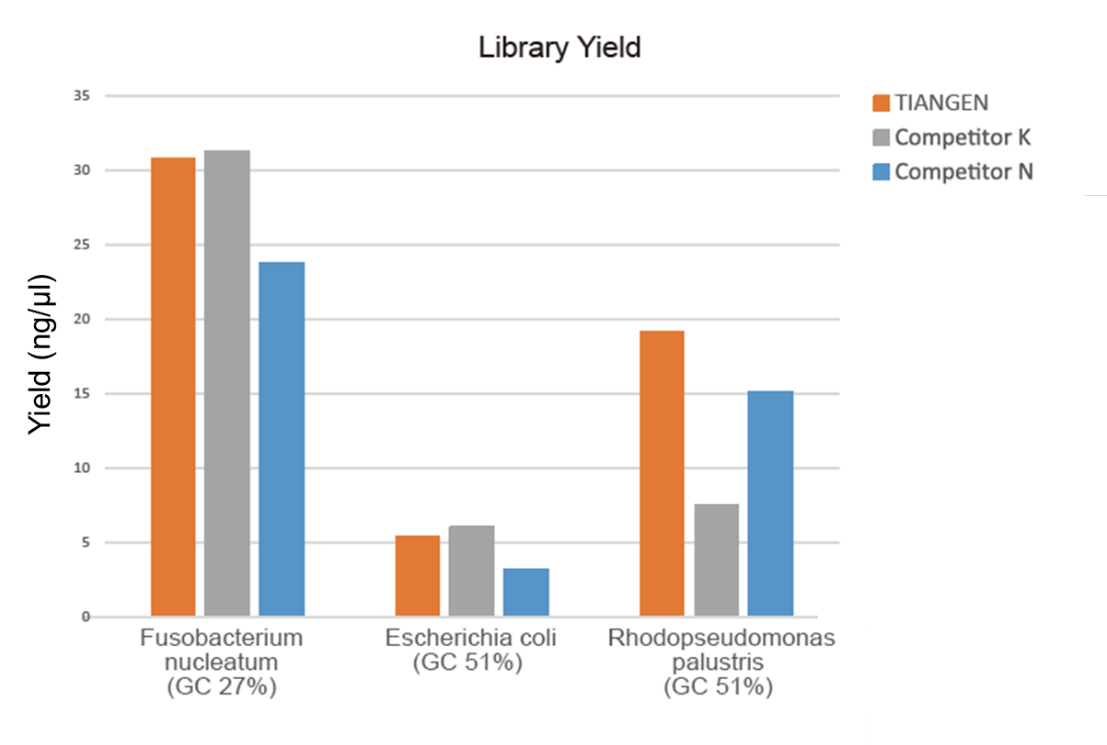 |
Larawan 1. Ang pagpapayaman ng library ng genomic DNA na may iba't ibang mga ratio ng GC (genomic input 10 ng, 8 cycle amplification) ay isinasagawa nang sabay-sabay sa pamamagitan ng paggamit ng TIANSeq HiFi Amplification Mix at HiFi enzyme mula sa Supplier K at N, at ang ani ng library ay napansin ng Agilent 2100. Ipinakita sa mga resulta na ang TIANSeq HiFi Amplification Mix ay may mataas na ani ng library, na may isang malakas na unibersalidad para sa iba't ibang mga nilalaman ng GC, at ang pagganap ng pagpapayaman ng library ay mas mahusay kaysa sa ibang mga tagapagtustos. |
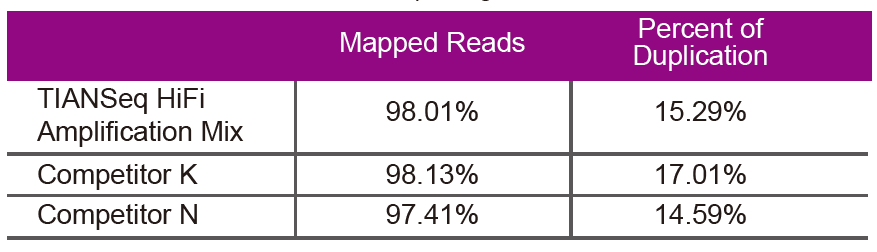 |
Data ng pagkakasunud-sunodGumamit ng TIANSeq HiFi Amplification Mix at HiFi enzyme na espesyal na ginagamit para sa pagpapalakas ng library ng NGS mula sa Supplier K at N para sa pagpapalakas ng library ng parehong genomic DNA (ang pag-input ng genome ay 10 ng). Pagkatapos ng pagkakasunud-sunod, pag-aralan ang saklaw at rate ng pagkopya ng library. |
Kagustuhan ng GC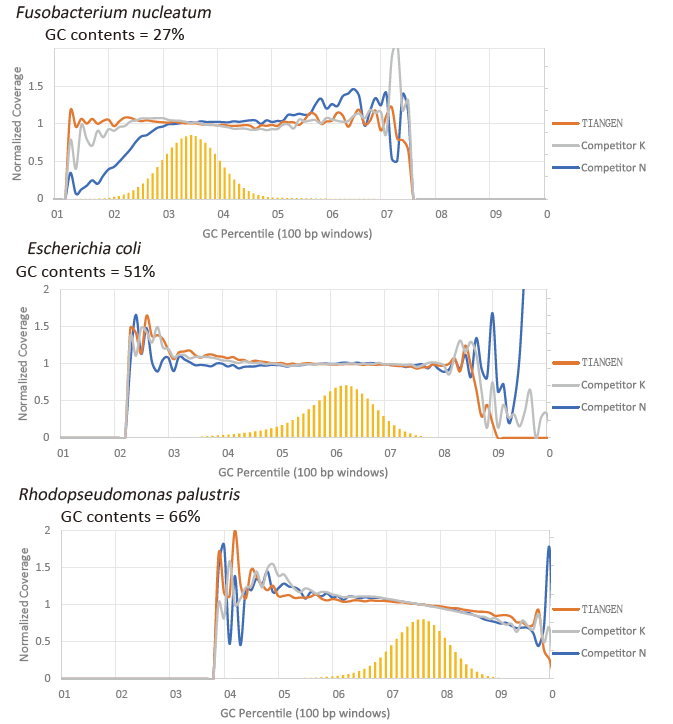 |
Larawan 2. Palakihin ang mga library ng genome na may iba't ibang mga nilalaman ng CG gamit ang TIANSeq HiFi Amplification Mix at ang HIFi mula sa Tagatustos K at N. Ipinapakita ng resulta na ang pagkakapareho ng TIANSeq HiFi Amplification Mix amplification library ay mabuti at walang kagustuhan ng GC, na katumbas ng mga resulta ng kumpanya K at bahagyang mas mahusay kaysa sa mga produkto ng kumpanya N. Ipinapakita ng mga resulta na ang saklaw ng TIANSeq HiFi Amplification Mix amplification library ay mataas, ang rate ng duplication ay natutugunan ang mga kinakailangan, at ang pagganap ng amplification ng library ay katumbas ng mga kakumpitensya. |
Sa kasalukuyan, ang teknolohiya ng pagsunud-sunod ng mataas na output ay pangunahing batay sa teknolohiyang pagsunud-sunod ng susunod na henerasyon. Dahil ang haba ng pagbabasa ng susunod na henerasyon na pagsunud-sunod ng teknolohiya ay limitado, dapat nating putulin ang buong pagkakasunud-sunod ng haba sa maliit na mga library ng fragment hanggang sa pagkakasunud-sunod. Ayon sa mga pangangailangan ng magkakaibang mga eksperimento sa pagsunud-sunod, karaniwang pinili namin ang solong-natapos na pagkakasunud-sunod o dobleng natapos na pagkakasunud-sunod. Sa kasalukuyan ang mga fragment ng DNA ng susunod na henerasyon ng pagkakasunud-sunod ng library ay karaniwang ipinamamahagi sa saklaw na 200-800 bp.

a) Ang DNA ay hindi maganda ang kalidad at naglalaman ng mga inhibitor. Gumamit ng mga de-kalidad na sample ng DNA upang maiwasan ang pagsugpo sa aktibidad ng enzyme.
b) Ang dami ng sample ng DNA ay hindi sapat kapag gumagamit ng PCR-free na pamamaraan upang mabuo ang DNA library. Kapag ang input ng fragmented na DNA ay lumampas sa 50 ng, ang PCR-free na daloy ng trabaho ay maaaring piliing isagawa sa panahon ng proseso ng pagtatayo ng library. Kung ang numero ng kopya ng silid-aklatan ay masyadong mababa upang direktang magkakasunud-sunod, ang library ng DNA ay maaaring mapalakas ng PCR pagkatapos ng adapter ligation.
c) Ang kontaminasyon ng RNA ay humahantong sa hindi tumpak na paunang pagsukat ng dami ng DNA Ang kontaminasyon ng RNA ay maaaring mayroon sa proseso ng paglilinis ng genomic DNA, na maaaring humantong sa hindi tumpak na dami ng DNA at hindi sapat na paglo-load ng DNA sa panahon ng pagtatayo ng silid-aklatan. Maaaring alisin ang RNA sa pamamagitan ng paggamot sa RNase.
A-1
a) Maliit na mga fragment (60 bp-120 bp) ay lilitaw Ang maliliit na mga fragment ay karaniwang mga fragment ng adapter o dimers na nabuo ng mga adapter. Ang paglilinis gamit ang Agencourt AMPure XP magnetic beads ay maaaring mabisang alisin ang mga fragment ng adapter at matiyak ang kalidad ng pagsunud-sunod.
b) Malalaking mga fragment ay lilitaw sa library pagkatapos ng PCR amplification Ang laki ng library ng fragment ng DNA ay tataas ng 120 bp pagkatapos ng adapter ay ligated. Kung ang fragment ng DNA ay tumataas ng higit sa 120 bp pagkatapos ng adapter ligation, maaaring sanhi ito ng hindi normal na fragment amplification ng labis na paglaki ng PCR. Ang pagbabawas ng bilang ng mga siklo ng PCR ay maaaring maiwasan ang sitwasyon.
c) Hindi normal na laki ng mga fragment ng DNA ng library pagkatapos ng ligation ng adapter Ang haba ng adapter sa kit na ito ay 60 bp. Kapag ang dalawang dulo ng fragment ay ligated sa mga adapter, ang haba ay tataas lamang ng 120 bp. Kapag gumagamit ng adapter maliban sa ibinigay ng kit na ito, mangyaring makipag-ugnay sa supplier upang magbigay ng nauugnay na impormasyon tulad ng haba ng adapter. Mangyaring tiyakin na ang daloy ng trabaho at pagpapatakbo ng eksperimento ay sumusunod sa mga hakbang na inilarawan sa manwal.
d) Hindi normal na laki ng fragment ng DNA bago ang adapter ligation Ang dahilan para sa problemang ito ay maaaring sanhi ng maling mga kundisyon ng reaksyon sa panahon ng fragmentation ng DNA. Ang iba't ibang mga oras ng reaksyon ay dapat gamitin para sa iba't ibang pag-input ng DNA. Kung ang input ng DNA ay higit sa 10 ng, inirerekumenda namin na piliin ang oras ng reaksyon ng 12 min bilang oras ng pagsisimula para sa pag-optimize, at ang laki ng fragment na ginawa sa oras na ito ay higit sa lahat sa saklaw na 300-500 bp. Maaaring dagdagan o bawasan ng mga gumagamit ang haba ng mga fragment ng DNA sa loob ng 2-4 min alinsunod sa kanilang sariling mga kinakailangan upang ma-optimize ang mga fragment ng DNA na may kinakailangang laki.
A-2
a) Ang oras ng pagkakawatak-watak ay hindi na-optimize Kung ang fragmented DNA ay masyadong maliit o masyadong malaki, mangyaring sumangguni sa Mga Alituntunin para sa Seleksyon ng Oras ng Fragmentation na ibinigay sa tagubilin upang matukoy ang oras ng reaksyon, at gamitin ang puntong ito bilang isang kontrol, bilang karagdagan, ang sistema ng reaksyon upang pahabain o paikliin ang 3 min upang makagawa ng mas tumpak na pagsasaayos sa oras ng pagkapira-piraso.
A-3
Hindi normal na pamamahagi ng sukat ng DNA pagkatapos ng paggamot ng pagkapira-piraso
a) Maling pamamaraan ng pagkatunaw ng fragmentation reagent, o ang reagent ay hindi ganap na halo-halong pagkatapos ng pagkatunaw. Matunaw ang 5 × Fragmentation Enzyme Mix reagent sa yelo. Kapag natunaw, ihalo nang pantay ang reagent sa pamamagitan ng dahan-dahang pagdulas sa ilalim ng tubo. Huwag vortex ang reagent!
b) Ang sample ng pag-input ng DNA ay naglalaman ng EDTA o iba pang mga pollutant Ang pag-ubos ng mga ion ng asin at chelating agents sa hakbang ng paglilinis ng DNA ay partikular na mahalaga para sa tagumpay ng eksperimento. Kung ang DNA ay natunaw sa 1 × TE, gamitin ang pamamaraan na ibinigay sa tagubilin upang magsagawa ng fragmentation. Kung ang konsentrasyon ng EDTA sa solusyon ay hindi sigurado, inirerekumenda na linisin ang DNA at matunaw ito sa deionized na tubig para sa kasunod na reaksyon.
c) Hindi tumpak na paunang pagbibilang ng DNA Ang laki ng pinaghiwalay na DNA ay malapit na nauugnay sa dami ng input ng DNA. Bago ang paggagamot ng fragmentation, ang tumpak na dami ng DNA gamit ang Qubit, Picogreen at iba pang mga pamamaraan ay mahalaga upang matukoy ang eksaktong dami ng DNA sa reaksyon ng system.
d) Ang paghahanda ng sistema ng reaksyon ay hindi sumusunod sa tagubilin Ang paghahanda ng fragmented na sistema ng reaksyon ay dapat na isagawa sa yelo nang mahigpit ayon sa mga tagubilin. Upang matiyak ang pinakamahusay na epekto, ang lahat ng mga bahagi ng reaksyon ay dapat ilagay sa yelo at ang paghahanda ng reaksyon ng sistema ay dapat na isagawa pagkatapos ng kumpletong paglamig. Matapos makumpleto ang paghahanda, mangyaring i-flick o pipet upang ihalo nang lubusan. Huwag vortex!
1. Ang hindi wastong pamamaraang paghahalo (vortex, marahas na oscillation, atbp.) Ay magdudulot ng abnormal na pamamahagi ng mga fragment ng silid-aklatan (tulad ng ipinakita sa sumusunod na pigura), kung kaya nakakaapekto sa kalidad ng silid-aklatan. Samakatuwid, kapag naghahanda ng solusyon sa reaksyon ng Fragmentation Mix, mangyaring marahan pipette pataas at pababa upang ihalo, o gamitin ang fingertip upang pumitik at ihalo nang pantay. Mag-ingat na hindi makihalo sa vortex.

2. Dapat gamitin ang mataas na kadalisayan ng DNA para sa pagtatayo ng silid-aklatan
■ Mahusay na integridad ng DNA: Ang electrophoresis band ay higit sa 30 kb, nang walang tailing
■ OD260 / 230:> 1.5
■ OD260 / 280: 1.7-1.9
3. Ang halaga ng pag-input ng DNA ay dapat na tumpak Iminungkahi na gumamit ng mga pamamaraan ng Qubit at PicoGreen upang sukatin ang DNA, sa halip na Nanodrop.
4. Ang nilalaman ng EDTA sa solusyon sa DNA ay dapat matukoy na ang EDTA ay may malaking impluwensya sa reaksyon ng fragmentation. Kung mataas ang nilalaman ng EDTA, kailangang gawin ang paglilinis ng DNA bago ang kasunod na pagsubok.
5. Ang solusyon sa reaksyon ng fragmentation ay dapat na ihanda sa yelo Ang proseso ng pagkakawatak-watak ay sensitibo sa temperatura ng reaksyon at oras (lalo na pagkatapos ng pagdaragdag ng enhancer). Upang matiyak ang kawastuhan ng oras ng reaksyon, mangyaring maghanda ng sistema ng reaksyon sa yelo.
6. Ang oras ng reaksyon ng pagkakawatak-watak ay dapat na tumpak Ang oras ng reaksyon ng pagkakahati-hati na hakbang ay direktang nakakaapekto sa laki ng mga produktong fragment, kung kaya nakakaapekto sa laki ng pamamahagi ng mga fragment ng DNA sa silid-aklatan.
1. Anong uri ng sample ang naaangkop sa kit na ito?
Ang naaangkop na uri ng sample ng kit na ito ay maaaring maging kabuuang RNA o purified mRNA na may mahusay na integridad ng RNA. Kung ang kabuuang RNA ay ginamit upang maitayo ang library, inirerekumenda na gamitin ang rRNA depletion kit (Cat # 4992363/4992364/4992391) upang alisin muna ang rRNA.
2. Maaari bang magamit ang mga sample ng FFPE upang makabuo ng library gamit ang kit na ito?
Ang mRNA sa mga sample ng FFPE ay mapapasama sa isang tiyak na lawak, na may medyo mahinang integridad. Kapag ginagamit ang kit na ito para sa pagtatayo ng silid-aklatan, inirerekumenda na i-optimize ang oras ng fragmentation (paikliin ang oras ng fragmentation o hindi gumaganap ng fragmentation).
3. Gamit ang hakbang sa pagpili ng laki na ibinigay sa manwal ng produkto, ano ang maaaring maging sanhi ng paglagay ng segment na lumilitaw ng bahagyang paglihis?
Isinasagawa ang pagpili ng laki sa mahigpit na alinsunod sa hakbang sa pagpili ng laki sa manwal ng produktong ito. Kung mayroong paglihis, ang dahilan ay maaaring ang mga magnetikong kuwintas ay hindi balanseng sa temperatura ng kuwarto o hindi ganap na halo-halong, ang pipette ay hindi tumpak o ang likido ay nanatili sa dulo. Inirerekumenda na gamitin ang mga tip na may mababang adsorption para sa eksperimento.
4. Pagpili ng mga adaptor sa pagtatayo ng silid-aklatan
Ang kit ng pagtatayo ng library ay hindi naglalaman ng reagent ng adapter, at inirerekumenda na gamitin ang kit na ito kasama ang TIANSeq Single-Index Adapter (Illumina) (4992641/4992642/4992378).
5. QC ng silid-aklatan
Detalye ng dami ng library: Ang Qubit at qPCR ay ginagamit upang matukoy ang konsentrasyon ng masa at konsentrasyon ng molar ng library ayon sa pagkakabanggit. Mahigpit na naaayon ang operasyon sa manwal ng produkto. Ang konsentrasyon ng silid-aklatan sa pangkalahatan ay makakamit ng mga kinakailangan ng pagsunud-sunod ng NGS. Pagtuklas ng saklaw ng pamamahagi ng library: Paggamit ng Agilent 2100 Bioanalyzer upang makita ang saklaw ng pamamahagi ng library.
6. Pagpili ng bilang ng ikot ng amplification
Ayon sa mga tagubilin, ang bilang ng mga PCR cycle ay 6-12, at ang bilang ng mga PCR cycle na kinakailangan ay dapat mapili alinsunod sa sample na input. Sa mga library na may mataas na ani, ang over amplification ay karaniwang nangyayari sa iba't ibang degree, na ipinakita ng isang maliit na mas malaking rurok matapos ang rurok ng target na saklaw sa pagtuklas ng Agilent 2100 Bioanalyzer, o ang napansin na konsentrasyon ng Qubit ay mas mababa kaysa sa qPCR. Ang banayad na paglaki ay isang normal na kababalaghan, na hindi nakakaapekto sa pagkakasunud-sunod ng library at kasunod na pagtatasa ng data.
7. Lumilitaw ang mga spike sa profile ng pagtuklas ng Agilent 2100 Bioanalyzer
Ang hitsura ng mga spike sa Agilent 2100 Bioanalyzer detection ay dahil sa hindi pantay na pagkakawatak-watak ng mga sample, kung saan magkakaroon ng mas maraming mga fragment sa ilang laki, at ito ay magiging mas halata pagkatapos ng pagpapayaman ng PCR. Sa kasong ito, iminungkahi na huwag gampanan ang pagpili ng laki, ibig sabihin itakda ang kondisyon ng pagkakawatak-watak sa 94 ° C sa loob ng 15 minutong nakapaloob, kung saan ang pamamahagi ng fragment ay maliit at puro, at ang homogeneity ay maaaring mapabuti.
Mga kategorya ng produkto
BAKIT MAPILI KAMI
Mula nang maitatag ito, ang aming pabrika ay nagkakaroon ng mga produktong pang-klase sa buong mundo sa pagsunod sa prinsipyo
ng kalidad muna. Ang aming mga produkto ay nakakuha ng mahusay na reputasyon sa industriya at valuabletrusty sa mga bago at lumang mga customer ..








