FastKing gDNA Dispelling RT SuperMix
Mga Tampok
■ Mabilis: Isang hakbang upang makumpleto ang pagtanggal ng genome at mahusay na reverse transcription sa loob ng 18 minuto sa pamamagitan lamang ng pagdaragdag ng mga template.
■ Mataas na kahusayan: Ang reverse transcriptase ay binago gamit ang isang hydrophobic motif, na may kahusayan sa RT na lumalagpas sa 95%.
■ Simple at madali: Ang eksklusibong thermosensitive DNase ay may mabilis na epekto, mataas na kahusayan na may mas maikli na oras ng reaksyon, at hindi makakaapekto sa cDNA.
Pagtutukoy
Uri: Binago ng Gene ang reverse transcriptase, gDNase
Mga Pamamaraan: Isang hakbang (pagtanggal ng genomic DNA at RT)
Kahusayan sa RT: > 95%
Template: 1 ng- 2 μg
Oras ng operasyon: ~ 18 min
Mga Aplikasyon: Ang reverse transcript cDNA ay maaaring magamit sa maginoo PCR, Real time PCR, pagtatayo ng library ng cDNA, SAGE (Serial Analysis of Gene Expression), primer extension at iba pang mga maginoo na eksperimento.
Ang lahat ng mga produkto ay maaaring ipasadya para sa ODM / OEM. Para sa mga detalye,mangyaring i-click ang Customized Service (ODM / OEM)
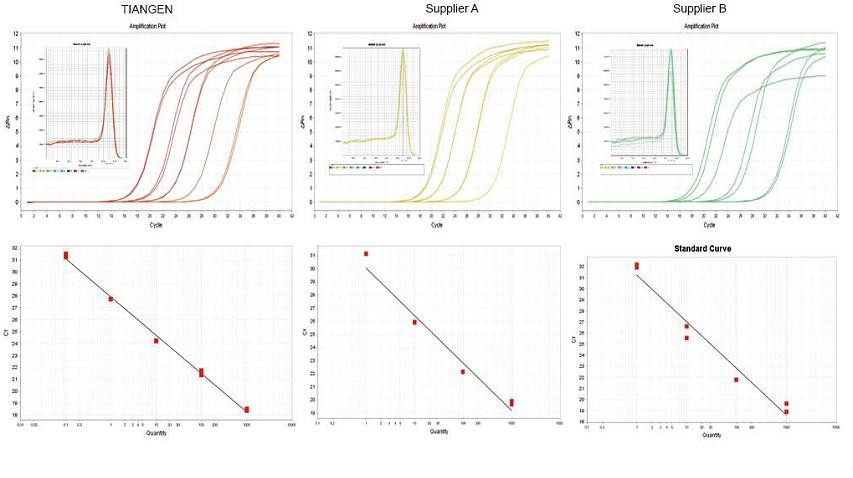 |
Pang-eksperimentong halimbawa 1. Ang cDNA ay na-synthesize gamit ang isang hakbang na pabalik na dami ng mga reagent ng TIANGEN FastKing gDNA Dispelling RT SuperMix, mga nauugnay na produkto mula sa Tagatustos A at Tagatustos B ayon sa pagkakabanggit. Nakita ang RN5 gene ng mga daga gamit ang TIANGEN Talent qPCR PreMix (SYBR Green), at ang amplve curve, melting curve at standard curve ay sinuri. Ipinapakita ng mga resulta na ang TIANGEN FastKing gDNA Dispelling RT SuperMix ay may pinakamataas na dami na Ct na halaga pagkatapos ng reverse transcription at mahusay na resistensya sa stress, at may halatang mga pakinabang para sa mga template na may mga residue na hindi malinis. |
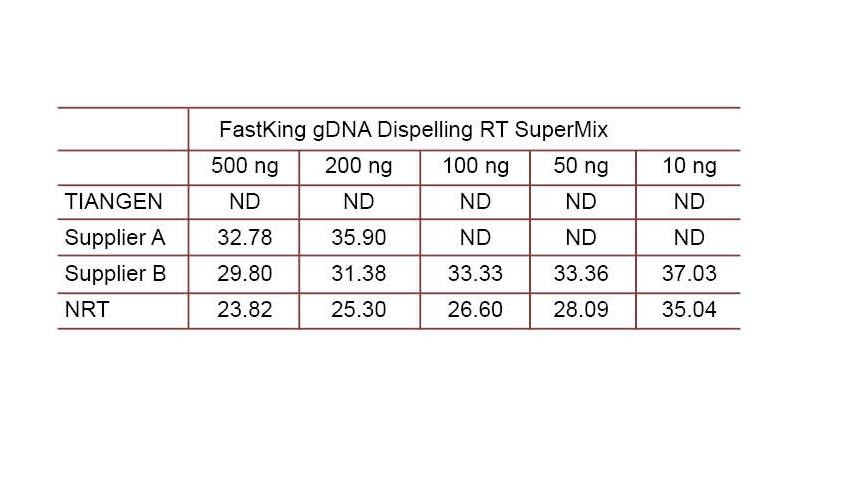 |
Pang-eksperimentong halimbawa 2. Ang cDNA ay na-synthesize gamit ang isang hakbang na pabalik na dami ng reagent mula sa TIANGEN FastKing gDNA Dispelling RT SuperMix, mga kaugnay na produkto mula sa Supplier A at Supplier B, ayon sa pagkakabanggit. Nakita ang gene ng tao na HsG gamit ang TIANGEN Talent qPCR PreMix (SYBR Green), at manu-manong magdagdag ng iba't ibang mga konsentrasyon ng genomic DNA upang makita ang kakayahang matanggal ang gDNA ng iba't ibang mga reagent. Ipinapakita ng mga resulta ng Ct na ang TIANGEN FastKing gDNA Dispelling RT SuperMix ay may mahusay na kakayahang alisin ang genomic DNA. Hanggang sa 500 ng ng labi ng genomic DNA ay maaaring matanggal nang perpekto nang hindi nakakaapekto sa mga resulta. ND: Hindi napansin. NRT: Pagtuklas ng halo nang walang reverse transcription. |
Ang A-1 RNA ay napahamak
—— Linisin ang de-kalidad na RNA na walang kontaminasyon. Ang materyal na kung saan nakuha ang RNA ay dapat na sariwa hangga't maaari upang maiwasan ang pagkasira ng RNA. Pag-aralan ang integridad ng RNA sa denatured gel bago ang reaksyon ng RT. Pagkatapos ng pagkuha ng RNA, dapat itong itago sa 100% formamide. Kung ginamit ang RNase inhibitor, ang temperatura ng pag-init ay dapat na <45 ° C, at ang pH ay dapat mas mababa sa 8.0, kung hindi man ay palabasin ng inhibitor ang lahat ng nakagapos na RNase. Bukod dito, ang RNase inhibitor ay dapat idagdag sa mga solusyon na naglalaman ng ≥ 0.8 mM DTT.
Naglalaman ang A-2 RNA ng mga inhibitor ng mga reaksyon ng reverse transcription
——Kasama sa mga inhibitor ng transcription sa SDS, EDTA, glycerol, sodium pyrophosphate, spermidine, formamide, guanidine salt, atbp. Paghaluin ang control RNA sa sample, at ihambing ang ani sa kontrol ng reaksyon ng RNA upang suriin kung mayroong isang inhibitor. Hugasan ang pag-ulan ng RNA na may 70% (v / v) na etanol upang alisin ang mga inhibitor.
A-3 Hindi sapat na pagsusubo ng mga primer na ginamit para sa pagbubuo ng unang strand ng cDNA
—— Tukuyin na ang temperatura ng pagsusubo ay angkop para sa mga primer na ginamit sa eksperimento. Para sa mga random hexamer, inirerekumenda na panatilihin ang temperatura sa 25 ° C sa loob ng 10 minuto bago maabot ang temperatura ng reaksyon. Para sa mga primer na partikular sa gene (GSP), subukan ang iba pang GSP, o lumipat sa oligo (dT) o random hexamer.
A-4 Maliit na halaga ng pagsisimula ng RNA
——Dagdagan ang dami ng RNA. Para sa mga sample ng RNA na mas mababa sa 50 ng, 0.1 μg hanggang 0.5 μg acetyl BSA ay maaaring magamit sa unang strand cDNA synthesis
A-5 Ang target na pagkakasunud-sunod ay hindi ipinahayag sa mga pinag-aralan na tisyu.
—— Subukan ang iba pang mga tisyu.
Nabigo ang reaksyon ng A-6 PCR
——Para sa dalawang hakbang na RT-PCR, ang template ng cDNA sa hakbang ng PCR ay hindi maaaring lumagpas sa 1/5 ng dami ng reaksyon.
A-1 Hindi tiyak na pagsusubo ng mga primer at template
——Ang 3'-end ng mga primer ay hindi dapat maglaman ng 2-3 dG o dC. Gumamit ng mga primer na partikular sa Gene sa unang synthesis ng strand sa halip na mga random primer o oligo (dT). Gumamit ng mas mataas na temperatura ng pagsusubo sa mga unang siklo, at pagkatapos ay isang mas mababang temperatura ng pagsusubo. Gumamit ng hot-start Taq DNA polymerase para sa PCR upang mapabuti ang pagiging tiyak ng reaksyon.
A-2 Hindi magandang disenyo ng mga primer na tukoy sa gen
—— Sundin ang parehong mga prinsipyo para sa disenyo ng panimulang amplification.
Ang A-3 RNA ay nahawahan ng genomic DNA
——Magamot ang RNA na may PCR-grade DNase I. Mag-set up ng isang reaksyon ng kontrol nang walang reverse transcription upang makita ang kontaminasyon ng DNA.
A-4 Bumubuo ng primer dimer
——Design ang mga primer nang walang mga komplimentaryong pagkakasunud-sunod sa 3 'end.
A-5 Masyadong mataas na Mg2+ konsentrasyon
—— I-optimize ang Mg2+ konsentrasyon para sa bawat template at kombinasyon ng primer
A-6 Nahawahan ng dayuhang DNA
—— Gumamit ng mga tip na lumalaban sa aerosol at mga enzyme na UDG.
A-1 Ang nilalaman ng unang produkto ng strand ay masyadong mataas
—— Bawasan ang dami ng unang produkto ng strand sa nakasanayang hakbang ng reaksyon ng PCR.
A-2 Masyadong mataas na halaga ng panimulang aklat sa reaksyon ng PCR
—— Bawasan ang input ng panimulang aklat.
A-3 Masyadong maraming mga cycle
——Optimize ang mga kundisyon ng reaksyon ng PCR at bawasan ang bilang ng ikot ng PCR.
A-4 Masyadong mababang temperatura ng pagsusubo
——Dagdagan ang temperatura ng pagsusubo upang maiwasan ang hindi partikular na pagsisimula at pagpapalawak.
A-5 Hindi tiyak na paglaki ng mga fragment ng oligonucleotide na nabuo ng pagkasira ng DNase ng DNA —— Kumuha ng mataas na kalidad na RNA upang maiwasan ang kontaminasyon ng DNA.
Ang RT-PCR ay upang baligtarin ang transcribe RNA sa cDNA, at pagkatapos ay gamitin ang reverse transcript cDNA bilang isang template para sa reaksyon ng PCR upang palakasin ang target na fragment. Pumili ng alinman sa mga random primer, Oligo dT at mga partikular na panimulang gen ayon sa mga tukoy na kundisyon ng eksperimento. Ang lahat ng mga primer sa itaas ay maaaring gamitin para sa maikling eukaryotic cell mRNA nang walang istraktura ng hairpin.
Random primer: Angkop para sa mahabang RNA na may istraktura ng hairpin, pati na rin ang lahat ng uri ng RNA tulad ng rRNA, mRNA, tRNA, atbp. Pangunahin itong ginagamit para sa reaksyon ng RT-PCR ng solong template.
Oligo dT: Angkop para sa RNA na may PolyA tailing (prokaryotic RNA, eukaryotic Oligo dT rRNA at tRNA ay walang PolyA tails). Dahil ang Oligo dT ay nakasalalay sa buntot ng PolyA, ang kalidad ng mga sample ng RNA ay kinakailangan upang maging mataas, at kahit na ang isang maliit na halaga ng pagkasira ay lubos na mabawasan ang dami ng buong-haba na synthesis ng cDNA.
Pambungad na tukoy sa Gene: Komplementaryo sa pagkakasunud-sunod ng template, na angkop para sa mga sitwasyon kung saan kilala ang pagkakasunud-sunod ng target.
Mayroong dalawang paraan:
1. Panloob na pamamaraan ng sanggunian: Sa teorya, ang cDNA ay mga fragment ng DNA na may iba't ibang haba, kaya't ang resulta ng electrophoresis ay pahid. Kung ang kasaganaan ng RNA ay mababa, walang produkto ang lalabas sa electrophoresis, ngunit hindi ito nangangahulugang walang produkto na mapalakas ng PCR. Sa pangkalahatan, ang panloob na sanggunian ay maaaring magamit upang makita ang cDNA. Kung ang panloob na sanggunian ay may mga resulta, ang kalidad ng cDNA ay maaaring garantisadong karaniwang (sa ilang mga kaso, kung ang target na fragment ng gene ay masyadong mahaba, maaaring may mga pagbubukod).
2. Kung may isang kilalang gene na pinalakas ng template na ito, maaari itong ma-verify ng mga panimula ng gen na ito. Ang pagpapalaki ng panloob na sanggunian ay hindi nangangahulugang walang problema sa cDNA. Dahil ang panloob na sanggunian ay may mataas na kasaganaan sa cDNA, madali itong palakasin. Kung ang cDNA ay bahagyang napinsala para sa iba't ibang mga kadahilanan, mula sa pananaw ng posibilidad, ang mga resulta ng PCR ng mababang kasaganaan na mga target na genes ay lubos na maaapektuhan. Habang ang panloob na sanggunian ay mataas pa rin sa kasaganaan, ang amplification ay marahil ay hindi maaapektuhan.
Bahagyang pagpapababa ng RNA. Nakita ang integridad at linisin ang RNA
Ang mga nilalaman ng RNA ng iba't ibang mga species ay maaaring magkakaiba, ngunit sa pangkalahatan, ang nakuha na kabuuang RNA ay dapat maglaman ng dalawang malinaw na 28S at 18S band sa gel electrophoresis, at ang ningning ng dating banda ay dapat na mas mataas nang dalawang beses kaysa sa huli. Ipinapahiwatig ng banda ng 5S na ang RNA ay napasama, at ang liwanag nito ay proporsyonal sa antas ng pagkasira. Ang matagumpay na pagpapalaki ng panloob na sanggunian ay hindi nangangahulugang walang problema sa RNA, dahil ang panloob na sanggunian ay nasa labis na kasaganaan, maaaring mapalakas ang RNA hangga't ang pagkasira ay hindi malubha. Ang OD260/ OD280ang ratio ng purong RNA na sinusukat ng spectrophotometer ay dapat na nasa pagitan ng 1.9 at 2.1. Ang isang maliit na halaga ng karumihan ng protina sa RNA ay magbabawas ng ratio. Hangga't ang halaga ay hindi masyadong mababa, ang RT ay hindi maaapektuhan. Ang pinakamahalaga para sa RT ay ang integridad ng RNA.
Ang pagpapalawak ng panloob na sanggunian na gene ay maaari lamang ipahiwatig na ang RT ay nagtagumpay, ngunit hindi ito kinakailangang nauugnay sa kalidad ng strand ng cDNA. Sapagkat ang panloob na mga fragment ng sanggunian sa pangkalahatan ay maliit sa laki at mataas sa pagpapahayag, mas madali silang maging matagumpay sa reverse transcription. Gayunpaman, ang laki at pagpapahayag ng target na gene ay nag-iiba mula sa gene hanggang sa gene. Ang kalidad ng cDNA ay hindi maaaring hatulan sa pamamagitan lamang ng panloob na sanggunian lalo na para sa mga target na fragment na mas mahaba sa 2 kb.
Ang ilang mga sample ay may kumplikadong pangalawang istraktura, o mayamang nilalaman ng GC, o mahalaga na may mababang kasaganaan. Sa mga kasong ito, dapat piliin ang naaangkop na reverse transcriptase alinsunod sa laki ng target na fragment at sample. Para sa mga template ng RNA na may mataas na nilalaman ng GC at kumplikadong pangalawang istraktura, mahirap buksan ang pangalawang istraktura sa mababang temperatura, o may karaniwang reverse transcriptase. Para sa mga template na ito, maaaring mapili ang Quant Reverse Transcriptase, dahil ang pagganap ng reverse transcription ay malinaw na mas mahusay kaysa sa serye ng M-MLV na reverse transcriptase, na maaaring baligtarin ang transcribe ng iba't ibang mga template ng RNA nang mahusay at isalin ang RNA sa unang strand ng cDNA sa maximum na lawak. Kapag gumagamit ng pangkalahatang reverse transcriptase kit, ang 20 μl na sistema ay maaari lamang mabisang baligtarin ang transcribe ng 1 μg ng kabuuang RNA. Mangyaring bigyang-pansin ang maximum na kapasidad ng RT ng kit. Kung ang template ay naidagdag nang labis, ang pabalik na transcription ay papabor sa RNA na may mataas na kasaganaan. Samakatuwid, mas mahusay na hindi lumampas sa maximum na kapasidad ng system.
A-1 Tukuyin kung ang RNA ay napasama nang malubha at kung ang RT ay matagumpay
Sa pangkalahatan, ang dahilan para sa kabiguan ng panloob na sanggunian na pagpapalaki ay madalas na sanhi ng malubhang pagkasira ng RNA. Ang isa pang posibleng dahilan ay kabiguan ng reverse transcription. Ang panloob na sanggunian ay hindi maaaring gamitin bilang isang pamantayan upang hatulan ang kalidad ng solong strand ng cDNA, ngunit maaari itong magamit bilang isang pamantayan upang hatulan kung matagumpay ang reverse transcription kung walang problema sa kalidad ng RNA. Ang pinakamahalagang bagay sa proseso ng reverse transcription ay upang mapanatili ang isang pare-pareho ang temperatura at isang pare-pareho na reaksyon ng system upang mapabuti ang kahusayan ng reaksyon.
A-2 Tukuyin kung ang mga panimula sa pagpapalaki ng mga panloob na sanggunian na gen ay maaasahan at kung mayroong anumang mga problema sa mga reagent na ginamit sa PCR.
Para sa kamag-anak na dami, ang RNA ay dapat na kwalipikado bago ang reverse transcription, na kinakailangan din sa maraming mga reverse transcription kit, halimbawa, bilangin ang input ng RNA bilang 1 μg. Dahil ang baligtad na transcript na cDNA ay isang halo-halong solusyon, kasama ang RNA, oligo dT, enzyme, dNTP, at kahit isang maliit na nalalabi sa DNA, dulot ng paglihis, kaya imposibleng tumpak na mabilang ang cDNA. Samakatuwid, kinakailangan ang dami ng RNA. Ang pagsasaalang-alang sa kahusayan ng reverse transcription ay pareho sa iba't ibang mga sample, ang dami ng nakuha na cDNA ay dapat na pareho, at ang dami na pagsusuri ay maaaring ipakita ang paghahambing ng mga antas ng pagpapahayag ng iba't ibang mga genes sa parehong halaga ng kabuuang RNA. Kapag nagsasagawa ng kamag-anak na fluorescence na dami ng PCR, ang dami ng cDNA ay maaaring hindi kailanganin pagkatapos ng reverse transcription dahil ang panloob na sanggunian na gene ay maaaring gampanan bilang sanggunian.
Pangunahing nauugnay ito sa mga gen, at ang reverse transcription ng mahabang fragment ay hindi magagawa para sa karamihan ng mga gen. Una, ang kahusayan ng reverse transcription ay mas mababa kaysa sa PCR. Pangalawa, ang rehiyon na mayaman ng GC at pangalawang istraktura ng maraming mga gen na nagbabawal sa parehong reverse transcription at PCR. Sa wakas, ang katapatan at amplification na kahusayan ng PCR ay mahirap garantiya nang sabay. Sa proseso ng reverse transcription, walang sinuman ang maaaring magagarantiyahan upang makakuha ng mahabang fragment para sa mga mababang kopya ng gen, lalo na ang paggamit ng oligo dT. Para sa 5 'UTR na may mas maraming GC, mas mahirap ito. Samakatuwid, ito ay pa rin isang makatuwirang pamamaraan upang baligtarin ang transcript na may mga random na primer, hanapin ang mga likas na lugar ng cleavage sa target na fragment, palakasin ng mga segment, at pagkatapos ay gawin ang paghihigpit sa pantunaw at ligation. Sa pangkalahatan, mahirap direktang palakasin ang mga fragment na mas malaki sa 2 kb, ngunit hindi palaging imposibleng makuha: 1. Una sa lahat, ginagarantiyahan ang integridad ng RNA / mRNA, at ang TRIZOL na pagkuha ay mas gusto. 2.M-MLV RT-PCR kit ay maaaring direktang magamit. Palawakin ang oras ng pagsusubo at taasan nang maayos ang bilang ng ikot sa proseso ng paglaki. Bilang kahalili, ang naka-salang PCR ay maaaring mailapat, o isakatuparan muna ang isa o dalawang reaksyon nang naaangkop na pinalawig na denaturation at extension time bago ang normal na paglaki ng PCR, na maaaring makatulong upang mapalawak ang mga fragment. Bigyang pansin ang katapatan ng polimerase. 3. Ang Long Taq ay maaaring magamit sa PCR upang makakuha ng mga perpektong resulta. 4. Para sa aplikasyon ng expression ng protina, dapat ilapat ang mataas na fidelity polymerase.
Mayroong dalawang uri ng reverse transcriptase na inaalok ng TIANGEN: Quant / King RTase at TIANScript M-MLV. Ang pangunahing pagkakaiba sa pagitan ng mga ito ay ang halaga ng pag-input ng mga template. Ang Quant ay isang natatanging reverse transcriptase, na naiiba mula sa karaniwang ginagamit na M-MLV na nagmula sa Moloney murine leukemia virus. Ang Quant ay isang bagong high-efficiency reverse transcriptase na muling ipinahayag ng engineering Escherichia coli. Ang dami ay angkop para sa amplifying 50 ng-2 μg ng RNA na may mataas na reverse transcriptal na aktibidad at mataas na ani. Kung ikukumpara sa ordinaryong MMLV o AMV, ang pinakamalaking katangian ng Quant ay mayroon itong napakalakas na pagkakaugnay sa mga template ng RNA at maaaring baligtarin ang mga kumplikadong template ng transcript nang walang mataas na temperatura na denaturation. Para sa mga template na may mas mataas na nilalaman ng GC, mas mataas ang pabalik na kahusayan. Gayunpaman, ang reverse transcriptase na ito ay may aktibidad na RNase H, na maaaring makaapekto sa haba ng produkto ng cDNA (angkop para sa <4.5 kb na mga template). Para sa maginoo na reverse transcription, inirerekumenda ang TIANScript MMLV reverse transcriptase. Ang RTase na ito ay binago na enzyme na may mahinang aktibidad na RNase H, na angkop para sa mahabang (> 5 kb) cDNA synthesis.
Ang isang hakbang na reverse transcription at paglaki ng PCR ay nakumpleto sa parehong tubo nang hindi binubuksan ang takip ng tubo sa pagitan ng cDNA synthesis at amplification, na kapaki-pakinabang upang mabawasan ang kontaminasyon. Dahil ang lahat ng mga halimbawa ng cDNA na nakuha ay ginagamit para sa pagpapalakas, ang pagkasensitibo ay mas mataas, na may minimum na 0.01 pg ng kabuuang RNA. Para sa matagumpay na isang hakbang na RTPCR, pangkalahatang ginamit ang mga primer na partikular sa gene upang simulan ang synthesidad ng cDNA. Ang dalawang hakbang na pamamaraan, katulad ng reverse transcription at PCR amplification ay isinasagawa sa dalawang hakbang. Isinagawa muna ang reverse transcription mula sa isang template ng RNA upang makakuha ng cDNA, at ang nakuha na cDNA ay napailalim sa isa o higit pang magkakaibang mga reaksyon ng PCR. Ang dalawang hakbang na pamamaraan ay maaaring gumamit ng oligo (dT) o mga random primer upang gabayan ang pagbubuo ng unang strand ng cDNA, at maaaring baligtarin ang transcribe ng lahat ng impormasyon ng mRNA mula sa isang tukoy na sample.
Mga kategorya ng produkto
BAKIT MAPILI KAMI
Mula nang maitatag ito, ang aming pabrika ay nagkakaroon ng mga produktong pang-klase sa buong mundo sa pagsunod sa prinsipyo
ng kalidad muna. Ang aming mga produkto ay nakakuha ng mahusay na reputasyon sa industriya at valuabletrusty sa mga bago at lumang mga customer ..